医歯薬学
2024.09.10
深層生成モデルを活用した一細胞レベルのmRNAスプライシングと分解の解析 ― 遺伝子発現制御メカニズムの解明に向けた革新的ツールの開発 ―
名古屋大学医学部附属病院卒後臨床研修・キャリア形成支援センターの水越周良研修医、国立がん研究センター研究所計算生命科学ユニットの小嶋泰弘独立ユニット長(東京医科歯科大学難治疾患研究所計算システム生物学分野連携研究員)、東京医科歯科大学難治疾患研究所計算システム生物学分野/名古屋大学大学院医学系研究科システム生物学分野の島村徹平教授らの研究グループは、各遺伝子の代謝を解析するための新規の情報解析手法「DeepKINET」を開発しました。
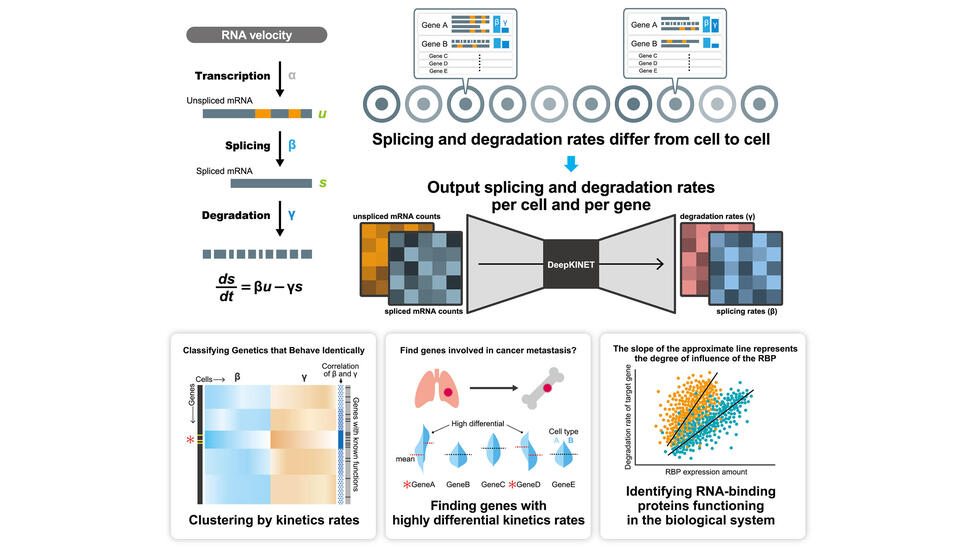
この手法は、一細胞トランスクリプトームデータと、RNA 速度モデル、深層生成モデルの枠組みを利用して、遺伝子のスプライシングと分解の速度を一細胞解像度で分析することを可能にする技術です。本解析手法を神経細胞、乳癌、骨髄異形成症状群のデータに適用し、各遺伝子の mRNA のスプライシングや分解を網羅的に解析することにより、RNA 結合タンパク質の機能や、スプライシング因子の変異の影響の推定を行いました。
mRNA のスプライシングや分解の理解は、遺伝子発現制御メカニズムの解明において不可欠であり、特にがんの発生や進行について新たな知見を提供します。DeepKINET は、一細胞トランスクリプトームデータを用いて、一細胞レベルで mRNA の動態を解析することに成功しました。本解析手法は、遺伝子発現制御の分子メカニズムに関する網羅的なデータに基づく仮説の提案を可能にし、新規の治療標的の探索に役立つと期待されます。
本研究成果は、国際学術誌 Genome Biology に 2024 年 9 月 6 日午前 1 時(英国夏時間)にオンライン掲載されました。
⚫ 一細胞トランスクリプトームデータから一細胞レベルで各遺伝子のスプライシング速度と分解速度の推定を行う手法、DeepKINET を開発。
⚫ RNA 結合タンパク質の機能やスプライシング因子の変異の影響の推定が可能。
⚫ 遺伝子発現制御メカニズムの解明により、創薬標的の探索や治療法開発への応用が期待。
◆詳細(プレスリリース本文)はこちら
掲載誌:
Genome Biology
論文タイトル: DeepKINET: a deep generative model for estimating single-cell RNA splicing and degradation rates
DOI: https://doi.org/10.1186/s13059-024-03367-8